1、Nature Biotechnology:AlphaFold2预测出98.5%的人类蛋白结构
2021年7月22日,来自英国DeepMind公司Demis Hassabis等研究人员合作在《自然》杂志上发表了题为“Highly accurate protein structure prediction for the human proteome”的研究成果,实现人类蛋白质组的高度精确蛋白质结构预测。
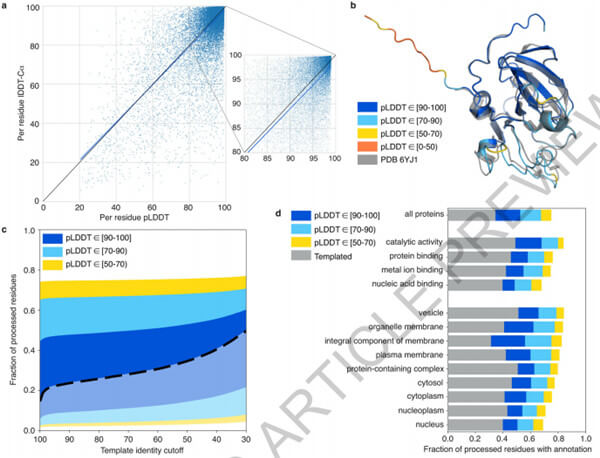
Fig 1|Model confidence and added coverage
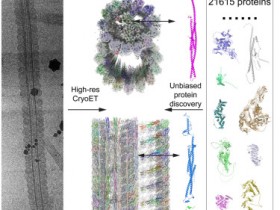
通过将最先进的机器学习方法AlphaFold2大规模地应用于几乎整个人类蛋白质组(98.5%的人类蛋白质),研究人员极大地扩展了结构的信息。由此产生的数据集涵盖了58%的有信心预测的残基,其中一个子集(占所有残基的36%)具有非常高的信心。研究人员介绍了在AlphaFold模型基础上开发的几个指标,并利用了这些指标来解释数据集,从而鉴定了强大的多结构域预测以及可能是无序的区域。最后,研究人员提供了一些案例研究,说明高质量的预测是如何被用来产生生物学假设的。
重要的是,研究人员通过一个公共数据库(由欧洲生物信息学研究所托管,网址:https://alphafold.ebi.ac.uk/)向社会免费提供这些预测结果。研究人员预计,常规的大规模和高精确度的结构预测将成为一个重要的工具,使新的问题能够从结构的角度得到解决。
据介绍,蛋白质结构可以提供宝贵的信息,既可用于推理生物过程,也可用于促成干预措施,如基于结构的药物开发或定向诱变。经过几十年的努力,人类蛋白质序列中的17%已被实验确定的结构所揭示。
(评论:大规模的准确结构预测将成为一种重要工具,让我们能从结构的角度解答新的科学问题,而AlphaFold的预测结果将帮助进一步阐明蛋白质的作用。)
文章来源:
Kathryn Tunyasuvunakool, Jonas Adler et al, Highly accurate protein structure prediction for the human proteome. DOI: 10.1038/s41587-021-00949-w, Nature Biotechnology:最新IF:31.864
2021年7月23日,来自美国耶鲁大学医学院Michael C. Crair研究组在《科学》杂志发表了题为“Retinal waves prime visual motion detection by simulating future optic flow.”的研究成果,发现视网膜波通过模拟未来光流来启动视觉运动检测。
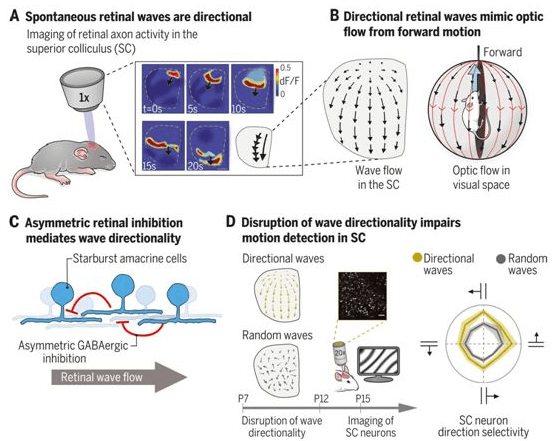
Fig 2|方向性视网膜波的起源和功能
他们描述了新生小鼠发育中的瞬态窗口,在此期间自发性视网膜波的空间传播类似于前向自运动产生的光流模式。他们表明波方向性需要形成成人方向选择性视网膜回路的相同回路组件,并且波方向性的慢性破坏会改变上丘神经元方向选择性反应的发展。这些数据展示了发展中的视觉系统如何模拟自发活动以模拟外部世界的行为学相关特征,从而指导自组织。
据了解,感知和响应环境刺激的能力出现在缺乏感官体验的情况下。睁眼前的自发性视网膜活动指导哺乳动物视网膜变位和眼睛特异性分离的改进,但其在高阶视觉反应特性发展中的作用仍不清楚。
(评论:深入了解在感觉经验开始之前调节大脑回路自我组织的活动依赖机制。)
文章来源:
Xinxin Ge, Kathy Zhang et al,Retinal waves prime visual motion detection by simulating future optic flow. DOI: 10.1126/science.abd0830 Science:最新IF:41.037
3、Nature Biotechnology:纳米孔直接RNA测序中识别差异化的RNA修饰新方法
2021年7月19日,来自新加坡A*STAR研究所Jonathan Göke等研究人员合作在知名学术期刊《自然—生物技术》发表了题为“Identification of differential RNA modifications from nanopore direct RNA sequencing with xPore.”的研究成果,他们开发出一项新技术,可从纳米孔直接RNA测序中识别差异化的RNA修饰。
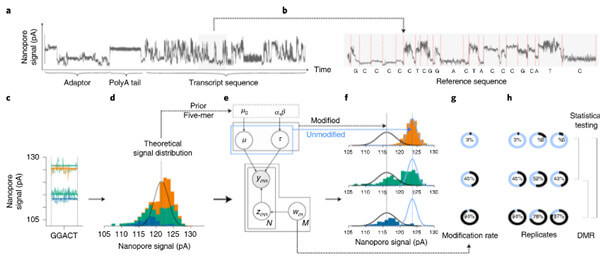
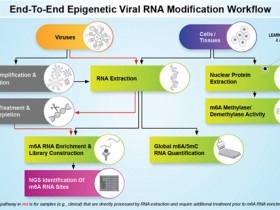
Fig 3| 原理流程
研究人员开发了一种计算方法,xPore,可从纳米孔直接RNA测序(RNA-seq)数据中识别不同的RNA修饰。研究人员在全转录组的N6-甲基腺苷(m6A)分析数据上评估了这个方法,从而证明xPore能以单碱基分辨率识别m6A位点的位置,估计细胞中被修饰的RNA物种的比例,并量化不同条件下的不同修饰率。
研究人员将xPore应用于六个细胞系和多发性骨髓瘤患者样本的直接RNA-seq数据,但没有匹配的对照样本,发现许多m6A位点在不同的细胞类型中都被保留下来,而有一个亚群在其修饰率上表现出显著的差异。这些结果表明,RNA修饰可以从直接RNA-seq数据中以较高的精度识别出来,从而能够从一个高通量实验中分析不同的修饰和表达。
(评论:RNA修饰(如m6A)调节了细胞RNA物种的功能。然而,量化RNA修饰的差异一直是个挑战。)
文章来源:
Ploy N. Pratanwanich, Fei Yao et al, Identification of differential RNA modifications from nanopore direct RNA sequencing with xPore. DOI: 10.1038/s41587-021-00949-w, 最新IF:31.864