近日,来自美国芝加哥大学Yingming Zhao和Lev Becker等研究人员在《Nature》上发表了提为“Metabolic regulation of gene expression by histone lactylation”的文章,发现组蛋白乳酸化修饰能够调控基因表达。
研究人员发现组蛋白赖氨酸残基的乳酸来源的乳酸化作为表观遗传修饰,直接促进了染色质的基因转录。研究人员在人类和小鼠细胞的核心组蛋白上鉴定出28个乳酸化位点。缺氧和细菌刺激通过糖酵解诱导乳酸的产生,并且其作为促进组蛋白乳酸化的前体。使用暴露于细菌的M1巨噬细胞作为模型系统,研究人员发现组蛋白乳酸化与乙酰化具有不同的时间动态。在M1巨噬细胞极化的后期,组蛋白的乳酸化修饰增强,从而诱导涉及伤口愈合的稳态基因,包括Arg1。
总体而言,这些结果表明,细菌攻击的M1巨噬细胞中的内源性“乳酸时钟”打开基因表达以促进体内平衡。因此,组蛋白乳酸化提供了一个增进了解乳酸的功能及其在各种病理生理状况(包括感染和癌症)中作用的机会。
据介绍,瓦博格效应(最初用于描述癌症中乳酸生成的增加)与多种细胞过程有关,例如血管生成、缺氧、巨噬细胞极化和T细胞活化。这种现象与多种疾病密切相关,包括瘤形成、败血症和自身免疫疾病。乳酸从肿瘤细胞中的丙酮酸转化而来,是众所周知的能源和代谢副产物。然而,其在生理和疾病中的非代谢功能仍然未知。
(评论:这也让我们了解到了癌细胞消耗营养物质的方式)
文章来源:
Di Zhang et al, Metabolic regulation of gene expression by histone lactylation,Nature,DOI: 10.1038/s41586-019-1678-1,最新IF:43.07
2、研究揭示琥珀酸受体结合拮抗剂的晶体结构
近日,来自瑞士诺华生物医学研究所Veli-Pekka Jaakola、Klemens Kaupmann和Matthias Haffke等研究人员在上发表了题为” Structural basis of species-selective antagonist binding to the succinate receptor”的文章,揭示了物种选择性拮抗剂与琥珀酸受体结合的结构基础。
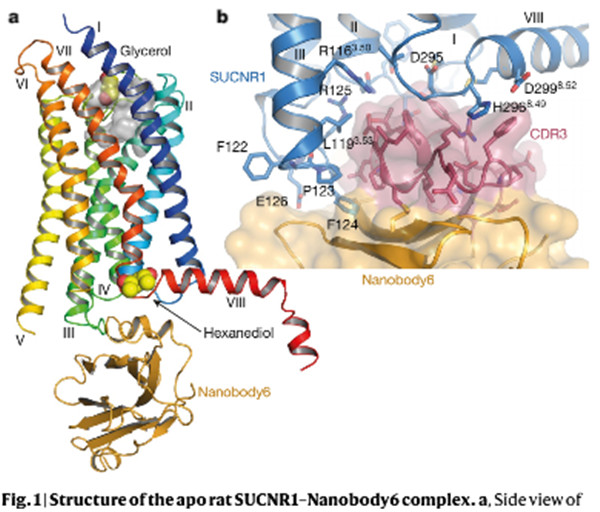
研究人员报道了大鼠SUCNR1与非活性构象的胞内结合纳米抗体形成复合物后的高分辨率晶体结构。基于结构的诱变和放射性配体结合研究与分子建模相结合,研究人员确定了物种选择性拮抗剂结合的关键残基,并能够确定人源化大鼠SUCNR1与高亲和力的、人类选择性拮抗剂NF-56-EJ40形成复合物后的高分辨率晶体结构。研究人员认为,这些关于琥珀受体的结构及其拮抗剂选择性的认知将促进基于结构的药物发现,并且将有助于进一步阐明SUCNR1在体外和体内的功能。
据悉,三羧酸循环中间体琥珀酸参与代谢过程,并在线粒体活性氧的稳态中扮演了至关重要的作用。负责琥珀酸信号传导的受体SUCNR1(也称为GPR91)是G蛋白偶联受体家族的成员,并将琥珀酸信号与肾素引起的高血压、视网膜血管生成和炎症联系在一起。由于SUCNR1将琥珀酸感觉为一种免疫危险信号,它与溃疡性结肠炎、肝纤维化、糖尿病和类风湿性关节炎有关,因此它作为治疗靶标引起了人们的关注。
(评论:学习了)
文章来源:Matthias Haffke et al, Structural basis of species-selective antagonist binding to the succinate receptor,Nature,DOI: 10.1038/s41586-019-1663-8, 最新IF:43.07
3、染色质拓扑结构的稳定保护基因组完整性
近日,来自丹麦哥本哈根大学Jiri Lukas和英国牛津大学Lothar Schermelleh等研究人员合作在Nature上发表了标题为“Stabilization of chromatin topology safeguards genome integrity”的论文,揭示染色质拓扑结构的稳定保证了基因组的完整性。
研究人员使用超分辨率显微镜显示53BP1和RIF1形成了一个自主的功能模块,其可稳定DNA断裂部位的三维染色质拓扑。此过程是通过将53BP1累积在紧密染色质的与拓扑相关域(TAD)序列共定位的区域中起始的,然后将RIF1募集到这些域之间的边界。
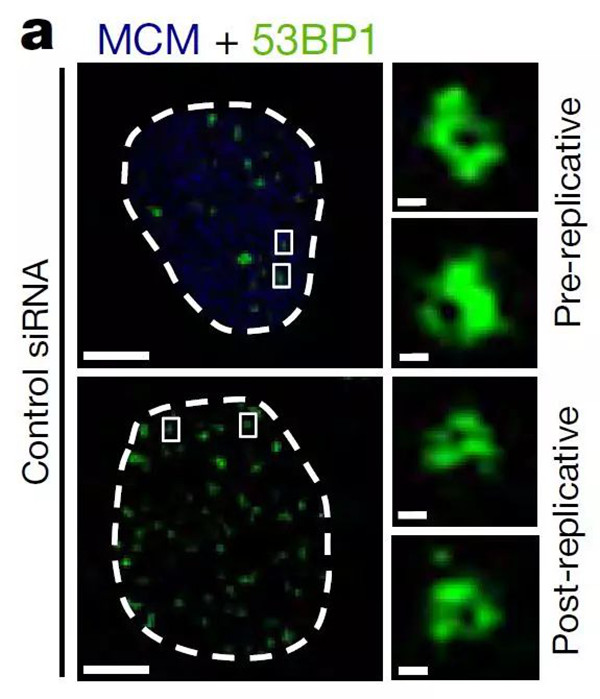
53BP1和RIF1的交替分布将单个DNA双链断裂(DSB)位点上几个相邻的TAD大小的结构稳定为有序的圆形排列。敲除53BP1或RIF1(而不是保护蛋白)会破坏这种排列方式,并导致DSB两边染色质失活、染色质间空间减少、DNA修复蛋白异常扩散以及DNA末端过度切除。cohesin蛋白的敲除会触发类似的拓扑畸变,这体现了DNA断裂后染色质结构的维持涉及塑造三维核组织的基本机制。
由于DSB两侧染色质的拓扑稳定与DNA修复无关,因此研究人员认为,除了提供结构支架以保护DNA末端免受异常加工之外,53BP1和RIF1还可以保护被DNA断裂所破坏的基因座的表观遗传完整性。
据介绍,为了保护DSB时基因组完整性,哺乳动物细胞动员相邻的染色质来保护DNA末端免受过度切除,这可能破坏修复保真度并损害健康染色体。53BP1负责这种形式的基因组监视,其在DSB处的积累触发了RIF1和shieldin-CST-POLα复合物的依次募集。这种途径如何反映和影响三维核结构尚不清楚。
(评论:学习了)
文章来源:Fena Ochs et al,Stabilization of chromatin topology safeguards genome integrity,Nature,DOI: 10.1038/s41586-019-1659-4, 最新IF:43.07