1、Nature:解析与端粒模板结合的人类CST-Polα-primase复合物结构
2022年7月13日,来自美国威斯康星大学麦迪逊分校Ci Ji Lim及其小组在学术期刊《自然》上发表了题为“Structures of the human CST-Polα–primase complex bound to telomere templates. ”的研究成果,解析与端粒模板结合的人类CST-Polα-primase复合物结构。
据介绍,哺乳动物的pol-α/primase对DNA代谢至关重要,它为若干DNA复制途径(如滞后链的合成和端粒C链的填充)提供新的RNA-DNA引物。Pol-α/引物酶如何单独或与附属蛋白合作,执行其复杂的多步骤引物合成功能的基本物理机制尚不清楚。
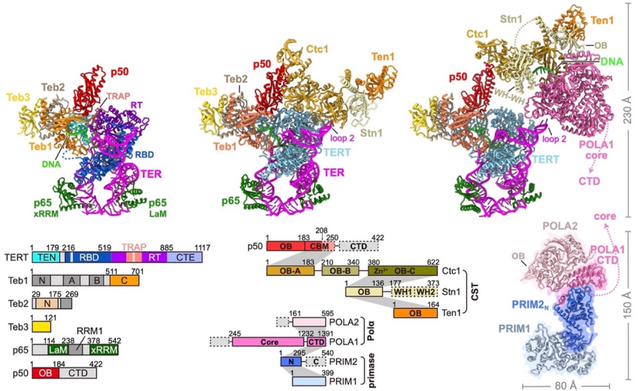
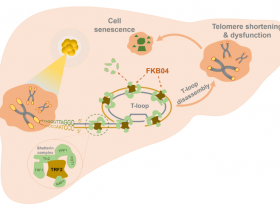
fig 1 | 四膜虫端粒酶 (左)、端粒酶–CST (中) 以及端粒酶–CST–PolαPrim (右) 的冷冻电镜结构。
研究人员发现,CST是DNA聚合酶-α/引物酶(pol-α/primase)的一个单链DNA结合的附属蛋白复合物,为高效的引物合成进行了物理设置。CST-pol-α/primase预起始复合物(PIC)与各种类型的端粒悬垂结合的冷冻电镜结构显示,与模板结合的CST将pol-α/primase的DNA和RNA催化中心分割成两个独立的域,并有效地将它们安排在RNA-DNA合成的顺序中。PIC架构为pol-α/primase RNA-DNA引物合成的多种结构需要提供了单一的解决方案。这项研究还揭示了对CST模板结合特异性、CST-pol-α/primase PIC装配的模板要求和激活的多种见解。
(评论:为理解端粒DNA G链和C链协同合成的分子机理提供了关键的结构基础)
文章来源:
He, Qixiang, Lin, Xiuhua et al, Structures of the human CST-Polα–primase complex bound to telomere templates.DOI: 10.1038/s41586-022-05040-1,Nature:最新IF:43.07
2、Nature:由CST蛋白复合物组织的端粒复制子的重建
2022年7月13日,来自美国科罗拉多大学博尔德分校Thomas R. Cech团队近期在《自然》杂志上发表了标题为“Reconstitution of a telomeric replicon organized by CST.”的研究论文,发现由CST蛋白复合物组织的端粒复制子的重建。
据介绍,端粒是线性染色体的自然末端,由重复DNA序列和相关蛋白质组成。端粒的复制可以使人类干细胞持续增殖,使癌细胞永生。这种复制需要端粒G链((TTAGGG)n)的单链DNA(ssDNA)的端粒酶延伸;互补C链((CCCTAA)n)合成的特征要少得多。CST(CTC1-STN1-TEN1)蛋白复合物是一种DNA聚合酶α-引物辅助因子,已知是体内端粒复制所必需的,本文的分子分析揭示了其机制的关键特征。
研究人员发现人类CST使用其ssDNA结合的活性来指定端粒C链合成的起源,并与Polα-primase结合。CST组织的DNA聚合可以复制折叠成G-四链体结构的端粒DNA模板,但该模板带来的挑战可能会导致体内观察到的端粒复制问题。结合端粒酶、短端粒ssDNA引物和CST-Polα-primase可提供完整的端粒DNA复制,从而产生与人类端粒上天然存在的相同类型的ssDNA 3'突出。研究人员得出结论,CST复合物不仅终止端粒酶延伸并将Polα-primase募集到端粒ssDNA,而且还协调C链的合成。由于端粒的复制具有与基因组其余部分的复制不同的特征,因此靶向包括CST在内的端粒复制成分有望用于癌症治疗。
(评论:为充分理解端粒DNA延长的分子机制提供了坚实的结构基础。)
文章来源:
Zaug, Arthur J., Goodrich, Karen J. et al, Reconstitution of a telomeric replicon organized by CST. DOI: 10.1038/s41586-022-04930-8, Nature:最新IF:43.07
3、Nature:核染色体所在的位置决定了分离错误的频率
2022年7月13日,来自荷兰Hubrecht研究所和乌得勒支大学医学中心Geert J. P. L. Kops团队在《自然》杂志上发表了标题为“Nuclear chromosome locations dictate segregation error frequencies.”的研究成果,发现核染色体所在的位置决定了分离错误的频率。
据了解,细胞分裂过程中的染色体分离错误会产生非整倍体和微核,它们会发生大量的染色体重排,如染色体碎裂。然后,选择性压力会形成不同的非整倍性和重排模式(例如在癌症中),但对于特定染色体是否存在分离错误和微核形成的初始偏差尚不清楚。
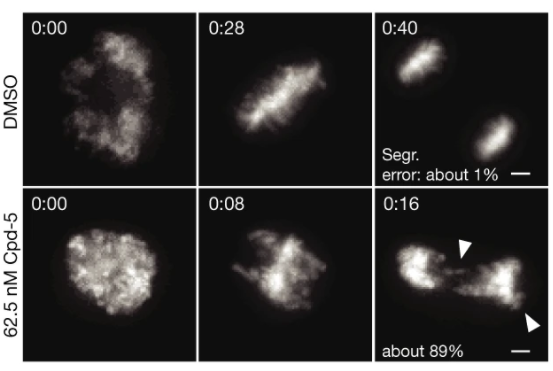
fig 3|有丝分裂的代表性静态图像。箭头表示错误分离的染色体
在未转化的二倍体细胞系和类器官中,使用易出错的有丝分裂后的单细胞DNA测序,研究人员发现染色体具有不同的分离错误频率,导致非随机的非整倍体景观。从这些细胞中分离和测序单个微核表明,错误分离的染色体也经常优先被包裹在微核中。在两种癌细胞系的天然存在的微核中也发现了类似的偏倚。
研究人员发现,单个染色体的分离错误频率与其在间期核中的位置相关,并表明在纺锤体两极后的外围染色体中是最高的。染色体位置的随机化Cas9介导的实时跟踪和单个染色体的强制重新定位表明,与核中心的距离越大,错误分离的倾向越大。因此,癌症基因组中的染色体碎裂和早期发育中的非整倍体更频繁地发生在较大的染色体上,这些染色体优先位于核外围附近。
(评论:研究结果揭示了核染色体位置、分离错误频率和微核含量之间的直接联系,这对大家理解肿瘤基因组进化和发育过程中特定非整倍体的起源具有重要意义。)
文章来源:
Klaasen, Sjoerd J., Truong et al, Nuclear chromosome locations dictate segregation error frequencies.DOI: 10.1038/s41586-022-04938-0, Nature:最新IF:43.07