做核酸-蛋白质互作的研究,心中没有乾坤,很容易无从下手,小编总结出核酸-蛋白质互作的常用研究方法,学霸们,get起来吧!
核酸-蛋白质的互作在生命活动中发挥着广泛而重要的作用, 二者的协作是各种生命现象的基础。将细胞或细胞器中的蛋白质、核酸等生物大分子的相互作用联系起来, 是综合研究一个完整的生物学途径的核心内容。常见的方法如下。
1、硝化纤维膜过滤实验
膜过滤方法在核酸-蛋白质复合物研究中渊源已久, 最初用于RNA-蛋白质的互作研究。其原理是蛋白与硝化纤维膜(Nitrocellulose filter, NCF)结合, 但DNA不与其结合。将标记过的DNA与蛋白质共同孵育, 用NCF过滤混合物, 则DNA能够通过NCF, 而DNA-pro-tein 复合物留在NCF上。随后干燥NCF, 通过标记物定量分析留在滤膜上的复合物, 即可判断二者的结合程度。
2、足迹法
足迹法最大的特点在于能确定蛋白结合DNA的片段长度, 其基本原理与DNA化学测序法相似, 首先将待测双链DNA片段进行标记, 然后加入适当浓度的探针对DNA进行消化剪切, 由于剪切具有随机性, 在反应完全时可将DNA切成单核苷酸。若控制探针浓度, 将DNA部分消化, 就可以形成一个单核苷酸、二核苷酸、三核苷酸……n核苷酸的混合物。经变性后电泳分离, 放射自显影, 即可形成以相差一个核苷酸为梯度的DNA条带。但当结合上蛋白时, 相应的DNA序列不会受探针的攻击, 因而在放射自显影图谱上DNA梯度条带在相应DNA结合蛋白的结合区域中断, 形成一空白区域, 恰似蛋白质在DNA上留下的足迹, 因而被形象地称作足迹法。
随着可选用的探针逐渐丰富,足迹法的具体研究方法和功能也丰富起来, 针对不同用途选取不同的探针, 在解决多种实际问题中起到了很大作用。足迹法又可细分为DNase I足迹法、外切酶Ⅲ足迹法、羟自由基足纹法、甲基化(DMS)保护/干扰实验、乙基化干涉实验等。
以上提到的多种足迹方法都可以给出核酸-蛋白复合体中核酸的长度或序列信息, 然而对于复合物中的蛋白组分的分析还需要结合其他的分析方法, 才能对核酸和蛋白质综合分析, 描述其全面的互作情况, 其中最为典型的方法是EMSA和Southwestern 杂交。
3、电泳迁移率变动实验(EMSA)
电泳迁移率变动实验又称为凝胶阻滞实验(Gel retardation assay)或凝胶移率变动实验(Gel mobility shift),是一种简单、快速和灵敏的用于体外检测蛋白粗提取物中特异性DNA结合蛋白的技术。
EMSA的原理是DNA片段在聚丙烯酰胺凝胶电泳中有一定迁移率, 而蛋白质结合后, 由于复合物在体积, 形状, 电荷以及结合位置的不同, 迁移率降低。传统方法用放射性标记的DNA探针(20~70 bp)与细胞提取物共孵育, 电泳后经放射自显影, 即可把与蛋白质结合的探针和自由DNA探针从电泳胶上分开。随着标记技术的发展, 地高辛、生物素等非放射性标记的采用, 避免了32P等标记探针有接触危险性且不容易定量分析的缺点。而且在此基础上发展起来的毛细管凝胶阻滞电泳技术, 结合了毛细管电泳的优势, 不需要使用放射性同位素, 样品用量少, 分辨率高, 解决了胚胎工程等研究中细胞数量限制的难题, 进一步完善了凝胶阻滞电泳的方法。
4、Southwestern杂交
DNA-蛋白质印迹杂交, 即Southwestern印迹杂交(Southwestern Blot)。将核蛋白粗提物进行SDS-PAGE电泳分析, 转膜后与同位素标记的特异DNA序列探针结合, 位点特异性DNA结合蛋白借助于氢键、离子键和疏水键结合DNA探针, 从而可通过放射自显影对DNA结合蛋白进行定性、定量分析。
5、染色质免疫沉淀(ChIP)
染色质免疫沉淀(Chromatin immunoprecipita-tion, ChIP)是体内研究蛋白质-DNA互作强有力的方法, 所谓体内(in vivo)方法, 是直接将完整的细胞进行交联处理, 保持其内核酸和蛋白质的结合情况, 之后再对核酸和结合蛋白做进一步分析。此方法不需要分别提取核酸或蛋白, 在天然的染色质环境下进行, 基本保持了二者在细胞内的真实结合状态。
ChIP方法最早广泛运用于酵母基因组的研究 中。在随后的十几年里, ChIP方法经过了研究者们不断的摸索和改进, 迅速发展。随着表观遗传学的兴起和发展, ChIP在染色体重塑的研究中崭露头角, 被运用于染色质相关的蛋白, 如组蛋白及其变异体, 特定DNA结构所结合的转录因子等研究中。ChIP的基本操作由对染色质的处理方法不同分为非变性和变性两种, 另外还根据不同实验需要产生了多种各具特色的方法, 如Re-ChIP、ChIP Chop等。一般进行完ChIP后直接用qPCR对DNA进行分析, 而近年来ChIP与芯片技术结合, 展现出更广泛的 用途。
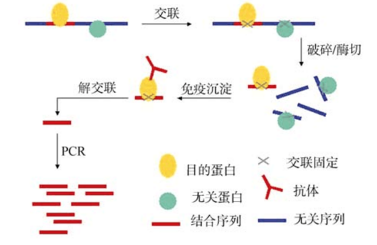
ChIP-on-chip是一种全基因组范围内的定位分析技术, 建立于染色质免疫沉淀(ChIP)和芯片技术(Chip)的联合运用之上。用于分析细胞中DNA结合蛋白对特异结合位点, 包括启动子、增强子、抑制子、沉默子、绝缘子、边界元件, 以及DNA复制的调控序列的鉴定, 整体研究生物体发育和病变过程中的复杂信息网络, 是一个绘制基因组功能元件作用网络的技术平台。
由于哺乳动物基因组的庞大和高重复性, 过去的研究一般都集中于对候选因子功能的确定而非全基因组的分析, 然而随着近年来Nimblegen等公司的哺乳动物基因组芯片的问世, 研究者不需要预先考虑蛋白质的可能结合位点, 对哺乳动物在全基因组水平上进行高分辨率的DNA结合蛋白, 染色质构成等研究也就成为可能。








