蛋白精氨酸甲基转移酶5(PRMT5)是与基因组组织、细胞周期调控和干细胞分化相关的关键调控蛋白。它与MEP50形成活性复合物,将甲基基团从S-腺苷甲硫氨酸(SAM)转移到组蛋白蛋白、转录因子和其他调控蛋白上。这些甲基化形成了单甲基化和对称二甲基化的精氨酸残基。PRMT5活性的失调与许多癌症有关,包括淋巴瘤、肺癌和乳腺癌。此外,PRMT5与肾脏疾病、心脏病和包括亨廷顿病和阿尔茨海默病在内的神经系统疾病有关。
最近的科学突破使PRMT5成为一个具有潜在药物靶点的有希望的新发现。对特异性抑制PRMT5和与其活性相关的代谢途径的新发现,引起了对这一重要蛋白的新关注。
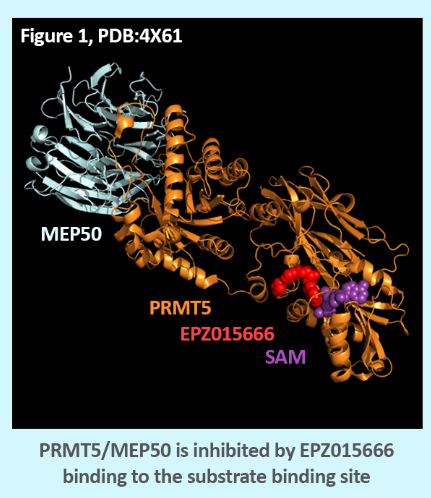
与所有酶靶标一样,一个主要挑战是找到不会显着抑制密切相关蛋白质的抑制化合物。简单的 SAM 类似物可以作为一般的甲基转移酶抑制剂,但不是特别特异。PRMT5 抑制的突破来自化学探针EPZ015666.EPZ015666 是通过竞争性抑制肽底物来抑制 PRMT5 活性的 nM 抑制剂(图 1)。此外,它具有选择性,对一组高达 mM 浓度的其他 PRMT 没有抑制作用。
除了直接抑制与蛋白质底物的相互作用外,通过靶向 PRMT5 对抗癌症的有趣策略还来自最近关于相关代谢途径的发现。甲基硫代腺苷磷酸化酶 (MTAP) 催化甲基硫代腺苷 (MTA) 的磷酸化,是甲硫氨酸补救途径的一部分.

编码 MTAP 的基因与肿瘤抑制基因 CDKN2A 密切相关(图 2)。这些基因位于 chr9p21 基因座,是多种癌症中常见的杂合缺失。最近关于 MTAP 在癌症中的作用的研究集中在 shRNA 筛选上,以鉴定对 MTAP 缺陷细胞增殖特别重要的基因.这些研究的共同点是观察到 MTAP ̄ 细胞对 PRMT5 和蛋氨酸腺苷转移酶 II α (MAT2A)(一种 SAM 生物合成途径酶)的依赖性显著增强。
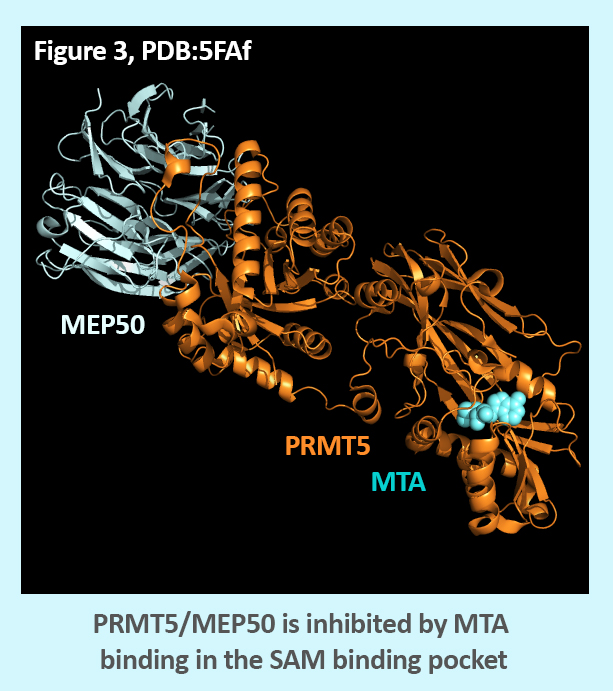
如图 2 所示,MTA 在 MTAP ̄ 细胞中积累。通过生化和结构研究,MTA 已被证明是 PRMT5 SAM 结合位点的竞争性抑制剂(图 3)MTA 对 PRMT5 也具有选择性。
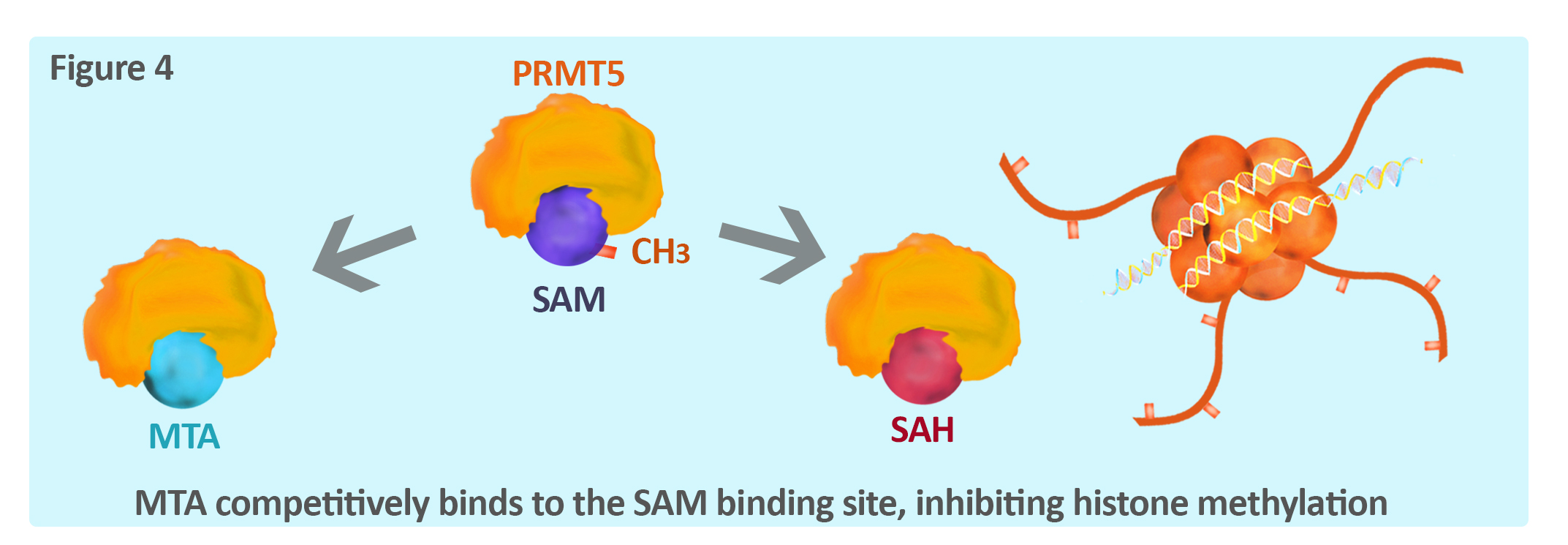
 这些研究表明,MTAP ̄ 细胞的 PRMT5 依赖性是由于 MTA 细胞水平升高部分抑制了 PRMT5 活性(图 4)。这为开发靶向 SAM 结合或稳定 MTA:PMRT5 复合物的 PRMT5 抑制剂提供了可能性。
这些研究表明,MTAP ̄ 细胞的 PRMT5 依赖性是由于 MTA 细胞水平升高部分抑制了 PRMT5 活性(图 4)。这为开发靶向 SAM 结合或稳定 MTA:PMRT5 复合物的 PRMT5 抑制剂提供了可能性。
MAT2A 在 SAM 生物合成中的作用及其与 MTAP 细胞增殖的联系也为基于 PRMT5 的癌症治疗提供了机会.MAT2A 的缺失已被证明会降低细胞 SAM 水平,并导致 MTAP ̄ 细胞中 PRMT5 活性的丧失(图 5)。这表明靶向 MAT2A 以降低 SAM 水平并打破有利于 MTA 抑制 MTAP ̄ 细胞中 PMRT5 的平衡的有趣可能性。

| 产品名称 | 货号 | 规格 |
| PRMT5 TR-FRET 检测试剂盒 | BPS-52171 | 384 reactions |
| PRMT5 化学发光检测试剂盒 | BPS-52073 | 384 rxns |
| PRMT5 化学发光检测试剂盒 | BPS-520021 | 96 rxns |
| PRMT5 均相检测试剂盒 | BPS-52052 | 384 reactions |
| MAT2A 抑制剂筛选检测试剂盒 | BPS-71402 |
|
| 甲基转移酶检测试剂盒 | BPS-52035 |
96 reactions
|
| PRMT5/MEP50,生物素标记 | BPS-52102 | 10 μg |
| PRMT5/MEP50、FLAG 标签、His 标签(Sf9 衍生) | BPS-51048 | 20ug |
BPS Bioscience 是值得信赖的 PRMT5 通路试剂、蛋白质、检测试剂盒和服务供应商和制造商。除了生物素标记的 PRMT5(BPS 货号 #52102)外,BPS 还提供 HEK293 衍生的 PRMT5 蛋白(BPS 货号 #51045)和 Sf9 衍生的 PRMT5 蛋白(BPS 货号 #51048)。PRMT5 相关蛋白包括 MTAP(BPS 货号 #50305)和 MAT2A(BPS 货号 #71401)。此外,BPS 还提供三种不同形式的 PRMT5 检测试剂盒;TR-FRET(BPS 货号 #52171)、均相(BPS 货号 #52052)和化学发光(96 次反应 [BPS 货号 #52002] 或 384 次反应 [BPS 货号 #52073])。
引文:
[1] Stopa, N., Krebs, J. E., and Shechter, D. (2015) The PRMT5 arginine methyltransferase: many roles in development, cancer and beyond, Cellular and Molecular Life Sciences : CMLS 72, 2041-2059.
[2] Chan-Penebre, E., Kuplast, K. G., Majer, C. R., Boriack-Sjodin, P. A., Wigle, T. J., Johnston, L. D., Rioux, N., Munchhof, M. J., Jin, L., Jacques, S. L., West, K. A., Lingaraj, T., Stickland, K., Ribich, S. A., Raimondi, A., Scott, M. P., Waters, N. J., Pollock, R. M., Smith, J. J., Barbash, O., Pappalardi, M., Ho, T. F., Nurse, K., Oza, K. P., Gallagher, K. T., Kruger, R., Moyer, M. P., Copeland, R. A., Chesworth, R., and Duncan, K. W. (2015) A selective inhibitor of PRMT5 with in vivo and in vitro potency in MCL models, Nature Chemical Biology 11, 432-437.
[3] Bertino, J. R., Waud, W. R., Parker, W. B., and Lubin, M. (2011) Targeting tumors that lack methylthioadenosine phosphorylase (MTAP) activity: current strategies, Cancer Biology & Therapy 11, 627-632.
[4] Kryukov, G. V., Wilson, F. H., Ruth, J. R., Paulk, J., Tsherniak, A., Marlow, S. E., Vazquez, F., Weir, B. A., Fitzgerald, M. E., Tanaka, M., Bielski, C. M., Scott, J. M., Dennis, C., Cowley, G. S., Boehm, J. S., Root, D. E., Golub, T. R., Clish, C. B., Bradner, J. E., Hahn, W. C., and Garraway, L. A. (2016) MTAP deletion confers enhanced dependency on the PRMT5 arginine methyltransferase in cancer cells, Science (New York, N.Y.) 351, 1214-1218.
[5] Marjon, K., Cameron, M. J., Quang, P., Clasquin, M. F., Mandley, E., Kunii, K., McVay, M., Choe, S., Kernytsky, A., Gross, S., Konteatis, Z., Murtie, J., Blake, M. L., Travins, J., Dorsch, M., Biller, S. A., and Marks, K. M. (2016) MTAP Deletions in Cancer Create Vulnerability to Targeting of the MAT2A/PRMT5/RIOK1 Axis, Cell Reports 15, 574-587.
[6] Mavrakis, K. J., McDonald, E. R., 3rd, Schlabach, M. R., Billy, E., Hoffman, G. R., deWeck, A., Ruddy, D. A., Venkatesan, K., Yu, J., McAllister, G., Stump, M., deBeaumont, R., Ho, S., Yue, Y., Liu, Y., Yan-Neale, Y., Yang, G., Lin, F., Yin, H., Gao, H., Kipp, D. R., Zhao, S., McNamara, J. T., Sprague, E. R., Zheng, B., Lin, Y., Cho, Y. S., Gu, J., Crawford, K., Ciccone, D., Vitari, A. C., Lai, A., Capka, V., Hurov, K., Porter, J. A., Tallarico, J., Mickanin, C., Lees, E., Pagliarini, R., Keen, N., Schmelzle, T., Hofmann, F., Stegmeier, F., and Sellers, W. R. (2016) Disordered methionine metabolism in MTAP/CDKN2A-deleted cancers leads to dependence on PRMT5, Science (New York, N.Y.) 351, 1208-1213.