对染色质三维结构的兴趣在过去的几年里发生了爆炸式的增长,我们现在知道,DNA并不是像一碗意大利面那样随意地堆积在细胞核中,而是按功能循环和结构域排列,更像是一个城市蓝图。
这对基因组科学家来说尤其令人兴奋,因为3D染色质体系结构可以帮助我们了解非编码单核苷酸多态性(SNPs)、DNA修饰和改变的蛋白质结合对远端(有时是染色体间基因)表达的影响。
第一个染色质构象捕获(3C) protocol于2002年发表,从那时起,新的染色质捕获技术的爆发令人震惊。如果你正在计划一个自己的3C实验,重要的是要了解主要的染色质捕获协议的异同点。
从本质上讲,所有的3C协议都是基于这样一种观点,末端的,功能相关的DNA片段更有可能相互作用。3C协议使用交联反应,通常是用1%的甲醛,以保持DNA片段和相关蛋白质之间的物理相互作用。接下来,染色质在预期的位置被消化,并带有限制性内切酶。然后,经消化的染色质被稀释并重新绑定。逆转交联和蛋白质消化后,形成一个混合DNA的库,如果片段在三维邻近,很有可能是结合在一起的。虽然大多数技术以这种标准协议的变化开始,但它们的输出范围和所能提供的答案各不相同。
一、染色质构象捕获(3 C)
最初的3C方法研究了DNA的两个预测部分之间的相互作用,这些“一对一”的假设是3C的亮点。当你想知道两个远端位点是否在三维原子核中相互作用时,这个技术是最好的。最初的3C使用精心设计的PCR引物来确定两个特定DNA片段之间的互动频率。
在3C可以测试两个基因组位点是否相互作用的情况下,4C实验的目标是找到多个与单一定位点相互作用的位点。反向引物是为单个片段设计的,这样PCR就会将所有与锚连接在一起的碎片进行循环和捕获。这种中等生产量的方法通常以测序结束,或者在阵列平台上运行结果库,以确定单个感兴趣位点的相互作用。
三、碳拷贝染色质构象捕获(5C)和HiC
最近,3C技术已被调整以适应更高的吞吐量实验。例如,5C的设计采用了多路复用引物和下一代测序(NGS)来确定在一个大的、预先确定的基因组区域内的许多相互作用,测定多点与多点的相互作用。这种类型的实验在描述一个潜在的监管区域的总体架构时最有用。
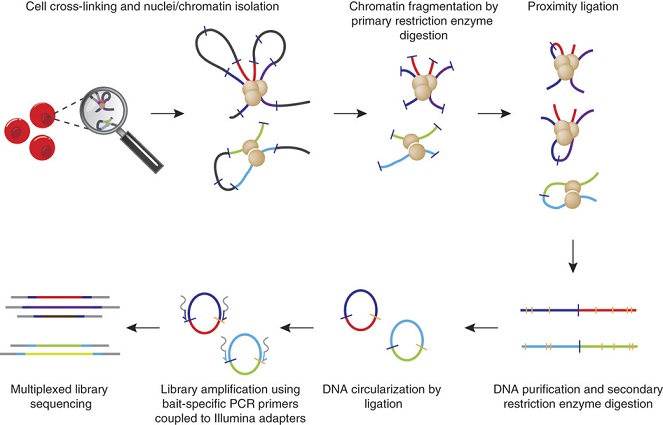
高通量染色体构象捕获(Hi-C)是在整个基因组水平上的功能,旨在测序和检测样品中所有可能的三维相互作用,实验流程如下。Hi-C大部分的努力都发生在生物信息学和解释的水平上,这些实验最常用于检查细胞在发育过程中或经过特定治疗后染色质景观的整体变化。
此外,越来越多的混合技术使用“C”方法为核心,例如,“Capture-HiC”使用DNA杂交探针从一个Hi-C库中拉下选定的目标位点。此技术特别擅长研究可能具有更高重要性的交互作用。类似地,ChIA-PET使用一种混合的染色质免疫沉淀/3C方法来研究DNA-蛋白质- DNA的相互作用,ChIA-PET在研究一种感兴趣的蛋白质是如何影响染色质结构时很有帮助。