1、Nature:绘制出原核基因的生物地理学图谱
2021年12月15日,来自复旦大学研究院、德国欧洲分子生物学实验室Peer Bork等研究人员合作在《自然》杂志上发表了题为“Towards the biogeography of prokaryotic genes.”的研究成果,该研究将地球上不同栖息地的微生物作为统一系统,运用人工智能技术对1.3万个公开宏基因组样本进行挖掘,构建了迄今为止最全面的全球微生物基因目录。
据介绍,微生物基因编码了地球上生命的大部分功能图谱。然而,尽管在各种栖息地的元基因组测序方面做出了越来越多的努力,但对全球生物圈的基因分布却知之甚少,这对人类和地球的健康有影响。
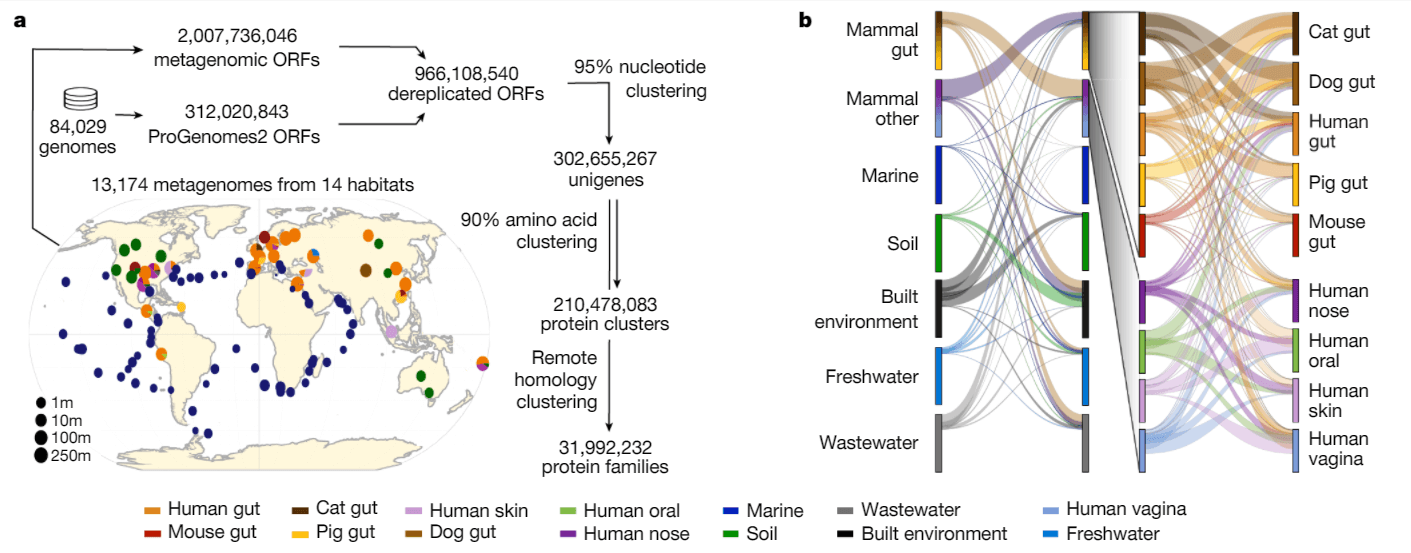
Fig 1 | Global Microbial Gene Catalogue(来源nature)
研究人员从14个主要栖息地的13174个公开的元基因组中构建了一个非冗余的基因目录,包括3.03亿个物种级别的基因(以95%的核苷酸身份聚类),并利用它来表明大多数基因是特定于单一栖息地的。在多个栖息地发现的一小部分基因富集在抗生素抗性基因和移动遗传元素的标记中。通过进一步将这些物种级基因聚类到3200万个蛋白家族中,研究人员观察到这些家族中的一小部分包含了大部分的基因(0.6%的家族占50%的基因)。大多数物种水平的基因和蛋白质家族是罕见的。此外,物种级基因,特别是罕见的基因,显示出较低的正向(适应性)选择率,从而支持了一种模型,即在每个蛋白质家族中观察到的大多数遗传变异性是中性或接近中性的。
(评论:构建了迄今为止最全面的全球微生物基因目录,对于理解微生物与人类健康的关系具有重要的作用。)
文章来源:
Coelho, Luis Pedro, Alves, Renato et al, Towards the biogeography of prokaryotic genes. DOI: 10.1038/s41586-021-04233-4, Nature:最新IF:43.07
2021年12月17日,来自北京大学张泽民等研究人员在《科学》杂志发表了标题为“Pan-cancer single-cell landscape of tumor-infiltrating T cells.”的研究成果。该研究绘制出肿瘤浸润T细胞的泛肿瘤单细胞图谱。
T细胞在癌症免疫治疗中发挥着核心作用,但人们缺乏对不同癌症类型的肿瘤浸润性T细胞的异质性和动态性的系统比较。

Fig 2 |21种癌症类型(来源Science截图)
研究人员为21种癌症类型的316名捐赠者建立了一个单细胞RNA测序的泛癌症T细胞图谱,癌症类型包括骨髓瘤、淋巴瘤、肾癌、卵巢癌、子宫内膜癌、食道癌、甲状腺癌、乳腺癌、胃癌和胰腺癌,并揭示了不同的T细胞组成模式。研究人员发现了CD8+T细胞耗竭的多种状态转换路径,以及这些路径在不同肿瘤类型中的偏好。某些T细胞群显示出与患者属性(如突变负担)的特定相关性,揭示了肿瘤微环境的可能决定因素。仅仅是肿瘤内的T细胞组成就可以将癌症患者划分为具有临床特征特异性的群体,为T细胞免疫和针对T细胞的精确免疫疗法提供了新的见解。
(评论:基于T细胞的肿瘤免疫分型为理解肿瘤浸润T细胞的总体特性提供了一个参考,也将进一步指导开发新的癌症免疫疗法和病人分层。)
文章来源:
Liangtao Zheng, Shishang Qin et al, Pan-cancer single-cell landscape of tumor-infiltrating T cells. DOI: abe6474 ,Science:最新IF:41.037
3、Nature Methods:实现小鼠全脑的跨模式相干配准
2021年12月9日,来自东南大学彭汉川课题组在《自然—方法学》杂志上发表了标题为“Cross-modal coherent registration of whole mouse brains.”的相关论文,实现小鼠全脑的跨模式相干配准。
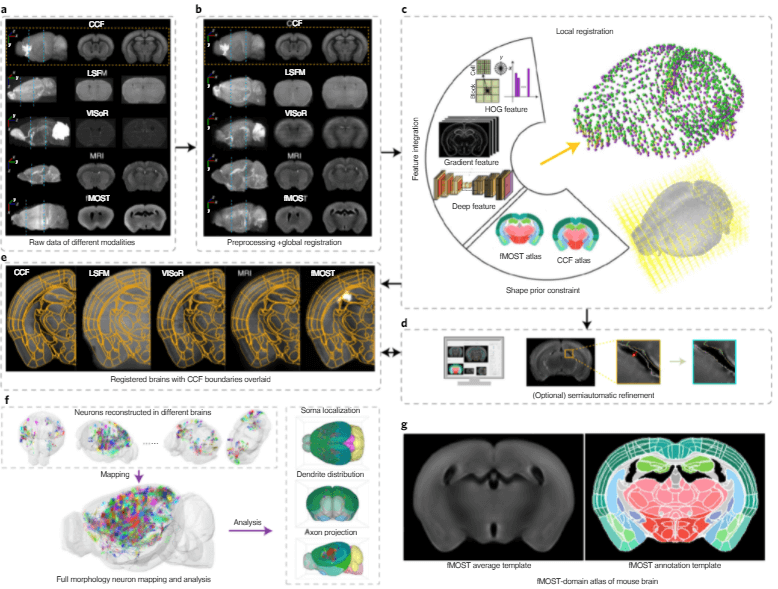
Fig 3| Overview of mBrainAligner(来源Nature Methods)
研究人员报道了一种跨模式的注册方法——mBrainAligner,它使用相干的地标映射和深度神经网络将小鼠全脑图像与标准的Allen共同坐标框架图集对齐。
该研究完成了31个高分辨率fMOST脑图像及1741个小鼠全脑神经元形态重建数据到Allen CCFv3图谱的映射,为BICCN细胞普查研究提供数据配准支撑;通过逆映射CCFv3构建了首个fMOST模态鼠脑图谱,展示了该图谱在生物和工程上的双重优势;给出了mBrainAligner在脑区划分、体细胞定位和轴突投射预测等任务以及STPT、fMOST、LSFM、VISoR和MRI不同模态脑图像间的交叉配准应用示例,以具有生物学意义的度量指标验证了其在跨模态脑图像配准中的优越性。
研究人员为荧光显微光学切片断层成像模式建立了一个脑图谱,以促进单细胞绘图,并使用这个方法生成了一个三维单神经元形态和神经元细胞类型的全脑图谱。
据了解,最近的全脑图谱项目正在收集大规模的三维图像,使用的方式包括:串行双光子断层扫描、荧光微光切片断层扫描、光片荧光显微镜、带有同步即时扫描和读出的容积成像或磁共振成像。将这些多维全脑图像注册到标准图谱上,对描述神经元类型和构建大脑线路图至关重要。然而,由于大脑解剖学的内在变化以及不同的样品制备方法和成像模式导致的伪影,跨模式的图像注册是具有挑战性的。
(评论:全脑神经元映射和解析是脑科学研究的重要方向,该研究mBrainAligner为多类模式生物脑图谱构建及相关神经生物学研究提供支撑)
文章来源:
Qu, Lei, Li, Yuanyuan, Xie, Peng et al ,Cross-modal coherent registration of whole mouse brains. DOI: 10.1038/s41592-021-01334-w. Nature Methods:最新IF:28.467