2025年3月24日,来自细胞生物学及生物物理学系Jan Ellenberg小组在《细胞》杂志发表了标题为“Nanoscale DNA tracing reveals the self-organization mechanism of mitotic chromosomes.”的研究成果,报道了纳米级DNA追踪技术揭示有丝分裂染色体自组织机制
据了解,基因组DNA如何在细胞分裂过程中折叠,形成具有特征的杆状有丝分裂染色体,这对忠实的基因组遗传至关重要,是生物学中一个长期悬而未决的问题。传统的观点认为,染色体内部可能存在一个像“骨架”一样的蛋白质支架,引导着DNA的有序排列。彻底颠覆了我们对染色体形成的认知。
这项最新的研究利用一种革命性的纳米级DNA追踪技术,以前所未有的清晰度,直接观察到了染色体形成的微观过程。令人惊讶的是,研究人员发现,染色体的棒状结构并非依赖于一个连续的“骨架”,而是通过一种更为动态和精巧的“自组织”机制实现的。
“分子拉链”:染色质环是如何形成的?
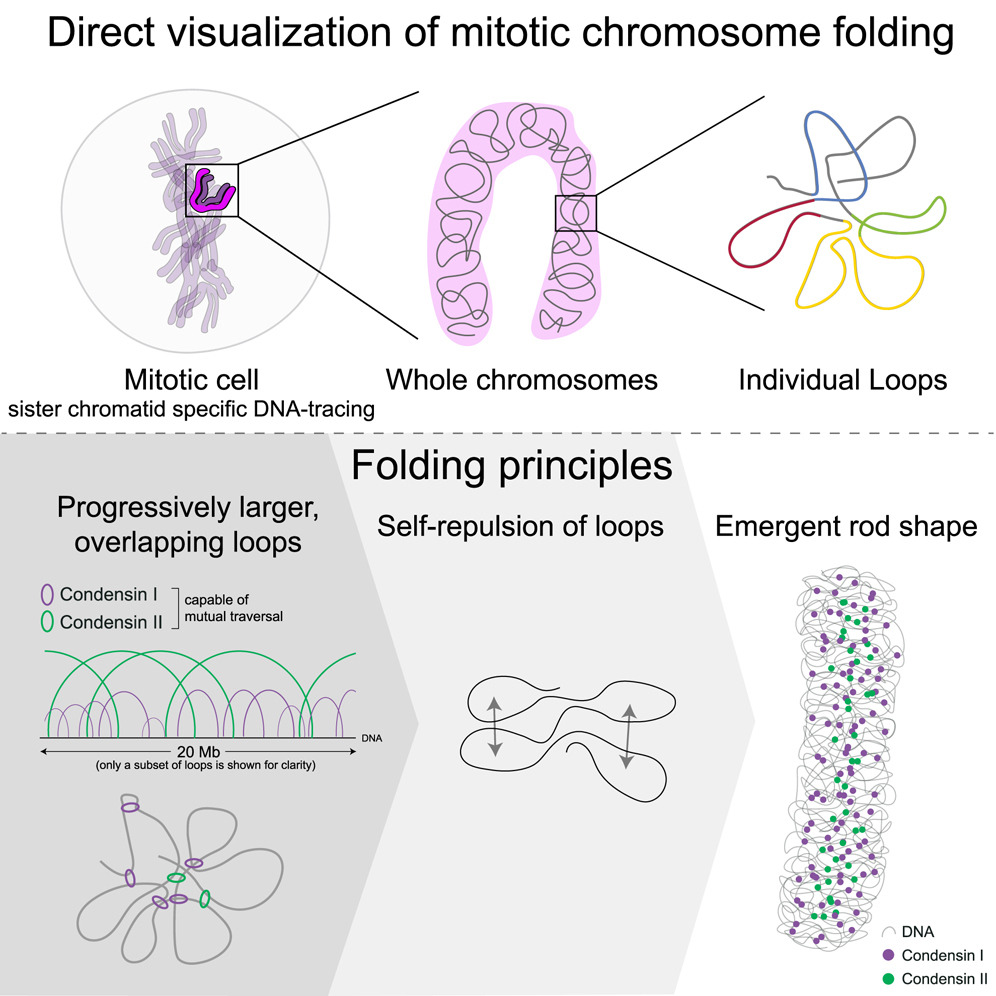
那么,染色体究竟是如何“自我组织”起来的呢?研究人员发现,染色体的形成关键在于一种叫做染色质环(DNA loop)的结构。想象一下,我们把一根长长的、柔软的绳子随意地弯曲,然后用一些小夹子在不同的位置将绳子的不同部分固定在一起,这样就形成了一个个的环状结构。染色体中的DNA就像这根绳子,而形成环的“夹子”则是一种特殊的蛋白质复合物,叫做凝聚蛋白(Condensin)。凝聚蛋白就像一个高效的“分子拉链”,它能够沿着DNA链移动,识别并结合DNA链上的特定位点,然后将DNA的不同片段连接在一起,从而形成一个个大小不一的环。这项研究发现,这些环的尺寸可以达到数百万个碱基对(megabases),这在整个基因组尺度上来说,是非常巨大的结构了。这项研究还进一步揭示,在染色体形成过程中,有两种主要的凝聚蛋白发挥着至关重要的作用:凝聚蛋白I(Condensin I)和凝聚蛋白II(Condensin II)。它们以一种协调的方式协同工作,驱动着DNA环的形成和扩展,就像两个熟练的工匠,共同塑造着染色体的基本结构单元。
环环相扣:重叠环的奇妙三维排列
更有趣的是,研究人员通过DNA追踪技术观察到,染色体中形成的这些DNA环并非彼此孤立和独立存在,而是以一种高度有序的方式相互重叠的。想象一下,我们用多根绳子在不同的位置打结,这些绳结可能会在三维空间中相互交织在一起,形成一个复杂的网络。染色体中的DNA环也是如此,它们在基因组上相互重叠,形成一种非常精密的、三维的网络结构。这种重叠的环结构对于染色体的进一步浓缩和形成其最终标志性的棒状形态至关重要。研究人员通过对不同染色体区域(例如第5号染色体、第2号染色体以及第14号染色体等)的细致分析,发现这种重叠环的形成模式在不同的染色体上是普遍存在的。他们还巧妙地利用不同尺度的DNA片段(例如1Mb、10Mb和100Mb)进行追踪,进一步证实了这种环的层级结构和普遍性。这种环环相扣、层层叠叠的排列方式,就像我们搭积木一样,通过不断地构建更高级别的结构,最终形成了我们在显微镜下看到的染色体。
“排斥力”显神威:染色体棒状结构的终极秘密
仅仅有DNA环的形成和重叠还不足以完全解释染色体最终呈现出的细长棒状结构。这项研究的另一个至关重要的发现是,这些相互重叠的DNA环之间存在一种微妙但强大的相互排斥的力量(self-repulsion)。想象一下,如果我们把很多带有相同电荷(比如都是负电荷)的小球放在非常近的地方,它们会因为同性电荷的排斥而相互推开,试图占据更大的空间。染色体中的DNA环也存在类似的现象,它们之间存在着一种物理上的排斥力,这种排斥力使得这些环在三维空间中不会仅仅是随机地缠绕在一起,而是会倾向于伸展开来,形成更加细长、规整的结构。为了验证这个想法,研究人员进行了巧妙的实验,他们对野生型细胞(WT cells)和经过TSA处理的细胞(TSA-treated cells)的染色体进行了长度和宽度测量。他们观察到,经过TSA处理后,染色体的长度会显著增加,而宽度则相应减小。TSA是一种组蛋白去乙酰化酶抑制剂(histone deacetylase inhibitor),它的作用是改变染色质的结构状态。
这些实验数据有力地表明,染色质的结构状态会影响DNA环之间的排斥力,从而最终影响染色体的整体形态。更令人信服的是,研究人员还进行了精密的计算机模拟,模拟了在有和没有这种环之间的排斥力的情况下,染色体的形成过程。模拟结果与他们在实验中观察到的真实的染色体形态高度吻合,这为环之间的自排斥是形成染色体棒状结构的关键驱动力提供了强有力的证据。他们甚至模拟了100Mb大小的染色体在不同条件下的形态,并将模拟结果与实验中观察到的2号染色体q臂(chr2 q-arm)的实际数据进行了细致的比较,结果显示了惊人的一致性。
纳米级追踪:以前所未有的精度揭示染色体组织的精细结构
这项研究之所以能够取得如此重要的、颠覆性的突破,很大程度上得益于其所采用的技术——纳米级DNA追踪技术。传统的光学显微镜技术虽然能够观察到染色体的整体形态,但在分辨染色体内部的精细结构方面存在着固有的局限性,例如很难清晰地分辨出单个DNA环的形状、大小和排列方式,以及它们之间的相互作用。而纳米级DNA追踪技术则能够以极高的分辨率,直接在纳米尺度上追踪单个DNA分子的路径。这就像给DNA分子装上了非常精确的“GPS”导航系统,让研究人员能够以前所未有的清晰度看到DNA在三维空间中的折叠方式和运动轨迹。通过这种先进的技术,研究人员得以直接观察到染色体中DNA环的形成、重叠以及它们之间微妙的相互作用,从而为我们揭示了染色体自组织机制的精细结构和动态过程。他们利用这种技术对不同染色体(例如第14号染色体)进行了深入细致的研究,并获得了大量的宝贵数据,这些数据为他们构建染色体形成的新模型提供了坚实而有力的实验证据。此外,研究人员还对经过TSA处理的细胞进行了类似的分析,通过比较处理前后染色体结构的变化,进一步验证了他们提出的自组织模型。
研究小组在单个分裂细胞中进行纳米级DNA追踪,以直接观察基因组DNA在有丝分裂期间从单环到整个染色体的三维折叠如何变化。他们的结构分析揭示了有丝分裂中具有6-8兆碱基的特征性基因组缩放。结合数据驱动模型和分子扰动,该研究团队可以表明,由凝聚形成的非常大且强烈重叠的环是有丝分裂染色体的基本结构原理。这些环在局部和全局上紧致染色体,达到染色质自排斥设定的极限。小组在3D中观察到的有丝分裂环的特征长度、密度和日益重叠的结构充分解释了细胞分裂过程中杆状有丝分裂染色体结构是如何通过自组织出现的。
文章来源:
Nanoscale DNA tracing reveals the self-organization mechanism of mitotic chromosomes
Author: Kai Sandvold Beckwith, Andreas Brunner et al, DOI: 10.1016/j.cell.2025.02.028,Cell:,最新IF:66.85







