1、Nature Methods: ZipSeq:一种利用光控空间编码进行实时单细胞空间转录组学研究的技术
2020年7月6日,来自美国加州大学旧金山分校Matthew F. Krummel课题组在《自然—方法学》上发表了题为 “ZipSeq: barcoding for real-time mapping of single cell transcriptomes. ”的研究成果,发展了一种利用光控空间编码进行实时单细胞空间转录组学研究的技术,称为ZipSeq。
课题组人员开发了名为“ZipSeq”的方法,通过特定模式照明和光笼寡核苷酸将DNA编码(‘zipcodes') 标记到完整组织的活细胞上,为后续单细胞组学数据记录细胞位置信息,具有实时和动态选择模式的特点。
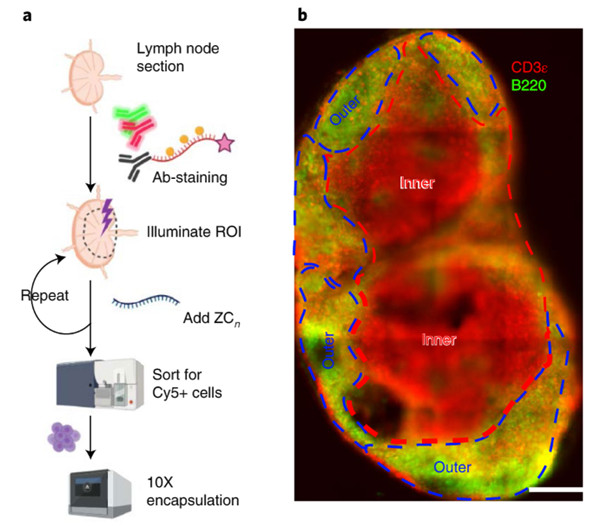
Fig 1 | ZipSeq mapping of single immune cell transcriptomes within a live lymph node section.
研究人员还利用ZipSeq对三个生物体系(体外伤口愈合、活淋巴结组织切片和活肿瘤微环境)的基因表达模式进行了映射。 在所有体系下,研究人员都发现了与组织结构相关联的新的基因表达新模式。在肿瘤微环境中,基因表达模式表明了骨髓和T细胞由外围向内的分化轨迹。ZipSeq的组合变体可以有效地缩放所定义区域的数量,从而为实时或微扰之后活组织基因表达模式的完整绘制提供了途径。
据介绍,空间转录组学技术需要同时获取单细胞转录组数据及其在多细胞生物中的三维空间位置信息。然而在活组织水平,将细胞位置与其单细胞转录组信息进行关联的现有方法仍存在多种限制。
(评论: ZipSeq作为一种新方法,增强了多细胞系统中空间异质性与组成细胞群体的转录异质性联系起来的能力。)
文章来源:
Kenneth H. Hu, John P. Eichorst et al, ZipSeq: barcoding for real-time mapping of single cell transcriptomes. DOI: 10.1038/s41592-020-0880-2. Nature Methods:最新IF:28.467
2、《自然—方法学》:单细胞数据中的高阶相互作用
2020年7月13日,来自澳大利亚悉尼大学的Jean Yee Hwa Yang课题组在《自然—方法学》上发表了题为“ Investigating higher-order interactions in single-cell data with scHOT. ”的研究论文。提供了一种单细胞数据高阶相互作用的研究算法。
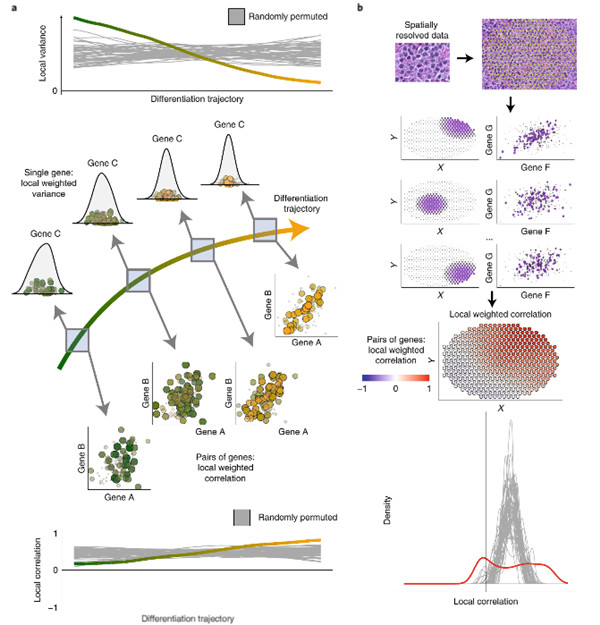
Fig. 2 | Methods workflow (来源 Nature Methods )
研究人员提供了一种灵活和统计学上稳健的用于确定基因中高阶相互作用的方法scHOT。scHOT可以应用于分析细胞的连续轨迹或跨空间信息,适用于包括变异性或相关性在内的各种高阶测量。小组通过研究小鼠肝脏胚胎发育过程中高阶相互作用的协同变化来展示了scHOT的使用。
此外,scHOT能够通过分析小鼠嗅球的空间转录组学数据识别空间上基因-基因相关性的微小变化。scHOT显著地改善了一阶微分表达式测试,并为从单细胞数据中挖掘高阶相互作用提供了框架。
据了解,单细胞基因组改变了科学家检查细胞命运选择的能力。研究细胞随着计算机有序“拟时间”的变化提供了解开关键基因在变异性和共变上微小变化的可能。
(评论: 能帮助科学家们利用先进的单细胞技术进行更为深入的研究)
文章来源:
Shila Ghazanfar, Yingxin Lin et al, Investigating higher-order interactions in single-cell data with scHOT. DOI: 10.1038/s41592-020-0885-x. Nature Methods:最新IF:28.467
3、Nature Methods:GPCR分子动力学模拟数据在线平台
2020年7月13日,来自西班牙Pompeu Fabra大学Jana Selent小组在《自然—方法学》上发表了题为“GPCRmd uncovers the dynamics of the 3D-GPCRome. ”的研究论文,建立了三维G-蛋白耦合受体(GPCRs)分子动力学模拟数据的在线共享平台。

Fig. 3: GPCRmd framework.(来源 Nature Methods)
这里研究人员建立了名为GPCRmd (http://gpcrmd.org/)的一个在线平台,整合了网络可视化功能以及全面、用户友好的分析工具箱。它允许来自不同学科的科学家可视化分析和共享GPCR分子动力学(MD)模拟数据。GPCRmd来源于领域内推动的创建一个开放的、交互式和标准化的GPCR分子模拟数据库的需求。GPCRmd是一个领域驱动的在线平台,用于可视化、分析和共享G-蛋白耦合受体(GPCR)分子动力学数据。该平台目前包含100%的GPCR类型,71%的受体亚型和80%的GPCR家族。
据了解,G-蛋白耦合受体(GPCRs)参与大量生理过程,是获批准药品最常见的靶点。在过去的十年里,GPCR的新的三维分子结构(3D-GPCRome)爆炸式增长,大大提高了针对这一蛋白家族的机理理解和药物设计机会。分子动力学(MD)模拟已经成为探索蛋白质原子水平构象广泛使用的技术。然而,分析和可视化分子动力学模拟数据需要有效的存储资源和专业软件。
(评论:学习了 )
文章来源:
Ismael Rodrguez-Espigares, Mariona Torrens-Fontanals et al , GPCRmd uncovers the dynamics of the 3D-GPCRome, DOI: 10.1038/s41592-020-0884-y. Nature Methods:最新IF:28.467