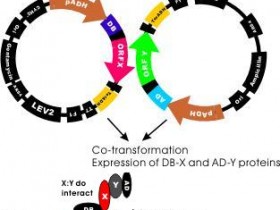
在前面的《关于酵母双杂交系统你需要知道这些》一文中我们提到“另外,在酵母双杂交的基础上,现在又发展出了酵母单杂交、酵母三杂交和酵母的反向杂交技术。它们被分别用于核酸和文库蛋白之间的研究、三种不同蛋白之间的互作研究和两种蛋白相互作用的结构和位点。”这回我们就来一一介绍酵母双杂交技术的兄弟姐妹们。
一、酵母单杂交技术
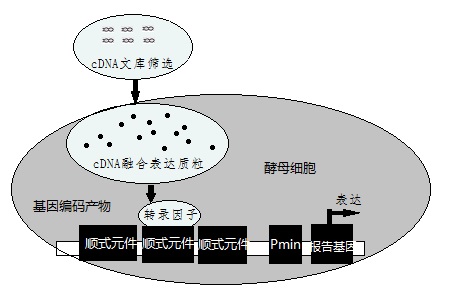
酵母单杂交原理示意图
酵母单杂交体系自1993年由Wang和Reed创立以来,在生物学研究领域中已经显示出巨大的威力。应用酵母单杂交体系已经验证了许多已知的DNA与蛋白质之间的相互作用,同时发现了新的DNA与蛋白质的相互作用,并由此找到了多种新的转录因子。酵母单杂交技术是一种研究蛋白质和特定DNA序列相互作用的技术方法,主要包括四个流程即:
1、筛选还有报告基因的酵母单细胞株;
2、构建表达文库;
3、重组质粒转化至酵母细胞;
4、阳性克隆菌株的筛选。
其基本原理为:真核生物基因的转录起始需转录因子参与,转录因子通常由一个DNA特异性结合功能域和一个或多个其他调控蛋白相互作用的激活功能域组成,即DNA结合结构域(DNA—bindingdomain,BD)和转录激活结构域(activationdomain,AD)。用于酵母单杂交系统的酵母GAL4蛋白是一种典型的转录因子,GAL4的DNA结合结构域靠近羧基端,含有几个锌指结构,可激活酵母半乳糖苷酶的上游激活位点(UAS),而转录激活结构域可与RNA聚合酶或转录因子TFIID相互作用,提高RNA聚合酶的活性。在这一过程中,DNA结合结构域和转录激活结构域可完全独立地发挥作用。据此,我们可将GAL4的DNA结合结构域置换为文库蛋白编码基因,只要其表达的蛋白能与目的基因相互作用,同样可通过转录激活结构域激活RNA聚合酶,启动下游报告基因的转录。
酵母单杂交技术的优缺点
随着酵母单杂交体系的不断发展和完善,它在科研、医疗等方面的应用将会越来越广泛,近来,已有应用酵母单杂交体系进行疾病诊断的研究报道。采用酵母单杂交体系能在一个简单实验过程中,识别与DNA特异结合的蛋白质,同时可直接从基因文库中找到编码蛋白的DNA序列,而无需分离纯化蛋白,实验简单易行。由于酵母单杂交体系检测到的与DNA结合的蛋白质是处于自然构象,克服了体外研究时蛋白质通常处于非自然构象的缺点,因而具有很高的灵敏性。目前,多种酵母单杂交体系的试剂盒和相应的cdna文库已经商品化,为酵母单杂交体系的使用提供了有利的条件。
但酵母单杂交也存在以下缺点:有时由于插入的靶元件与酵母内源转录激活因子可能发生相互作用,或插入的靶元件不需要转录激活因子就可以激活报告基因的转录,因此往往产生假阳性结果。如果酵母表达的AD融合蛋白对细胞有毒性,或融合蛋白在宿主细胞内不能稳定地表达,或融合蛋白发生错误折叠,或者不能定位于酵母细胞核内,以及融合的Gal4AD封闭了蛋白质上与DNA相互作用的位点,则都可能干扰AD融合蛋白结合于靶元件的能力,从而产生假阴性结果。
酵母单杂交的基本操作过程
(1)设计含目的基因(称为诱饵)和下游报告基因的质粒,并将其转入酵母细胞。
(2)将文库蛋白的编码基因片段与GAL4转录激活域融合表达的cDNA文库质粒转化人同一酵母中。
(3)若文库蛋白与目的基因相互作用,可通过报告基因的表达将文库蛋白的编码基因筛选出来。在这里作为诱饵的目的基因就是启动子DNA片段,文库基因所编码的蛋白就是启动子基因结合蛋白。
酵母单杂交技术的用途
迄今为止,应用酵母单杂交体系已经识别并验证了许多与目的DNA序列结合的蛋白质,同时单杂交技术还被应用于识别金属反应结合因子。正向与反向单杂交体系的结合,还可用于筛选阻碍DNA与蛋白质相互作用的突变的单个核苷酸。
目前,在研究DNA—蛋白质相互作用中,酵母单杂交体系主要有以下3种用途:
(1)确定已知DNA—蛋白质之间是否存在相互作用
(2)分离结合于目的顺式调控元件或其他短DNA结合位点蛋白的新基因
(3)定位已经证实的具有相互作用的DNA结合蛋白的DNA结合结构域,以及准确定位与DNA结合的核苷酸序列。
二、酵母三杂交技术
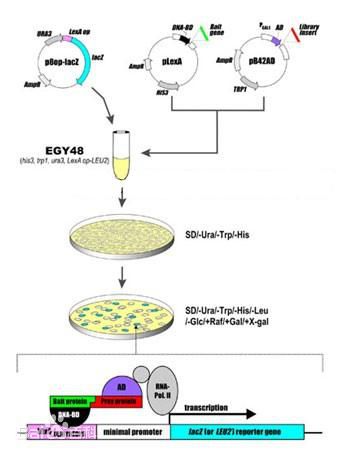
酵母三杂交系统(three-binding hybrid system)用于分析蛋白和RNA间的相互作用。三杂交系统提供了快速、多用的体内检测RNA-蛋白间相互作用的新方法。与双杂交系统相比,杂交RNA与杂交蛋白有些不同:首先,杂交RNA分子可能不是生理结构,杂交的不同RNA组分对正常结构会有一定影响。但Dhruba的研究表明,局部、相对稳定的结构区如MS2 coat protein,IRP1识别位点等可在体内共存于同一杂交RNA分子中。其次,这些RNA中的茎环区结构相当稳定,RNA只要形成部分正确结构,则足以介导转录激活作用。Dhruba等在IRP1与IRE的RNA相互作用的研究中,第一个RNA结合蛋白选用噬菌体MS2被壳蛋白(MS2 coat protein),MS2 coat protein可识别RNA中的21nt的茎环序列,并与之有较高的亲和力。将其与LexA融合构建杂合蛋白。IRP1与Gal4的激活结构域(activation domain)融合构建另一个杂合蛋白。IRE mRNA在非编码区有21nt的茎环区序列,编码区编码与IRP1特异性结合的蛋白序列。在实验中他们用一质粒表达杂交RNA,含2个MS2 coat protein结合位点和1个IRE。
三杂交系统的基本原理是将一个已知的RNA结合蛋白与转录因子的DNA结合domain(如LexA)构建第一个融合蛋白,第二种蛋白(待选的RNA结合蛋白)与转录激活结构域构建融合蛋白。此外构建和表达一杂合RNA,含有二个不同的结合位点。当RNA与二个RNA结合蛋白的结合位点相互作用时可激活报道基因的转录和表达。
Senguptaxi等首先报道了应用三杂交系统分析金属调节蛋白(iron regulatory protein-1,IRP1)与金属反应元件(iron response element,IRE)RNA序列,以及HIV转录激活蛋白(trans-activator protein, Tat)与HIV转录激活反应元件(HIV trans-activation response element,TAR)RNA序列间的作用。
象双杂交系统一样,三杂交系统具有广泛的应用。
(1)应用此系统可分析确定RNA--蛋白作用的精确结构域,甚至单个核苷酸或氨基酸残基。
(2)用于鉴定和克隆识别结合有重要生理功能RNA(如转录、定位、RNA病毒包装和感染等)的蛋白质,可用于调控的机理研究、疾病的防治以及抗病毒药物的研制开发。
(3)过构建杂交RNA库,可筛选与特定蛋白结合的RNA。
(4)有可能在此基础上发展四杂交系统研究RNA-RNA间的相互作用。
三、酵母的反向杂交技术
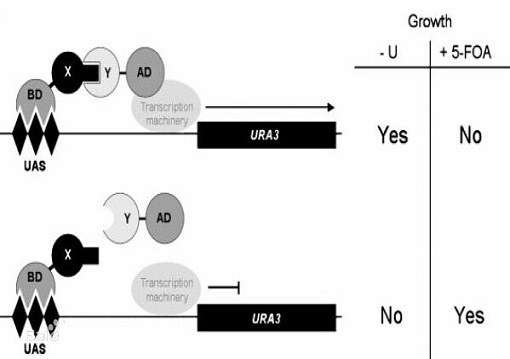
确定了蛋白之间的相互作用后, 更重要的工作是研究其功能、结构及其作用的条件调节。鉴定在相互作用的一对蛋白中的任一蛋白突变对其相互作用的抑制作用, 不仅有助于探索相互作用的结构单位, 而且可作为研究体内功能的遗传学方法。这对于多个作用分子的蛋白就更为重要。 1996年vidal酵母双杂交系统基础上建立了反向酵母双杂交系统(reverse two-hybrid system,又称逆双杂交系统), 提供了简便的鉴定阻断蛋白间相互作用的方法。
反向酵母双杂交系统的关键是掺入一种表达产物对细胞生长有毒的报导基因, 用于监测蛋白间的相互作用。例如酵母ura3基因表达产物是尿嘧啶合成所必需的, 同时它又可催化5-氟乳清酸(5-foa)转化为有毒物质。vidal等构建了一酵母细胞株, 其ura3的表达由含gal4结合位点的启动子严密控制。此细胞株在缺乏尿嘧啶的培养基中培育需要gal4激活结构域(gad)和gal4 dna结合结构域(gbd)的融合蛋白的相互作用的表达。而在含5-foa的完全培养基中则受gad和gbd融合蛋白相互作用的抑制。因此可以用5-foa抗性克隆从随机突变库中鉴定阻断蛋白相互作用的突变体。
反向双杂交系统可更有效地蛋白间作用的关键位点或起决定作用的个别氨基酸, 进而分析蛋白结构和功能的关系。此外此系统还可筛选能阻止某些蛋白间相互作用的肽或小分子物质用作临床治疗制剂。