GSCAlite由来自华中科技大学生命科学院郭安源教授课题组开发,提供了一系列服务来执行基因组学(Expression, SNV, CNV and methylation)和免疫基因组(24个免疫细胞)分析。此外,结合临床信息和小分子药物,用户可以挖掘候选生物标志物和有价值的小药物,以进行更好的实验设计和进一步的临床试验。GSCA整合了来自TCGA的33种癌症类型和超过750种来自GDSC和CTRP的小分子药物的一万个基因组数据。通过ImmuCellAI对24个免疫细胞进行了免疫基因组分析。
其中GSCA的4个主要功能模块分别是Expression、Immune、Mutation、Drug, 大家所感兴趣的差异表达、GSEA、生存分析、GSVA、免疫细胞浸润、CNV、DNA突变、DNA甲基化、药物敏感性,以及上述多种因素间的关联分析,它统统能做!工具地址戳这里:http://bioinfo.life.hust.edu.cn/GSCA/
今天就以其中的Mutations模块做介绍,其他的三个模块大家可探索一番,Mutations模块涉及的内容包括SNV、CNV、甲基化,看下方介绍:
1、比如针对(PRAD)前列腺腺癌,我们输入感兴趣的基因ACTB, AR, FN1, FOS, JUN, MYL9,勾选SNV这部分就可以展示大家输入的基因的突变数目、突变频率、突变类型、突变对生存的影响(包括OS和PFS)。这部分里有漂亮的火柴图用来展示每个基因上各个突变位点的突变率,有bar plot和oncoplot展示不同突变类型的比例和不同基因的突变频数频率。如下图1
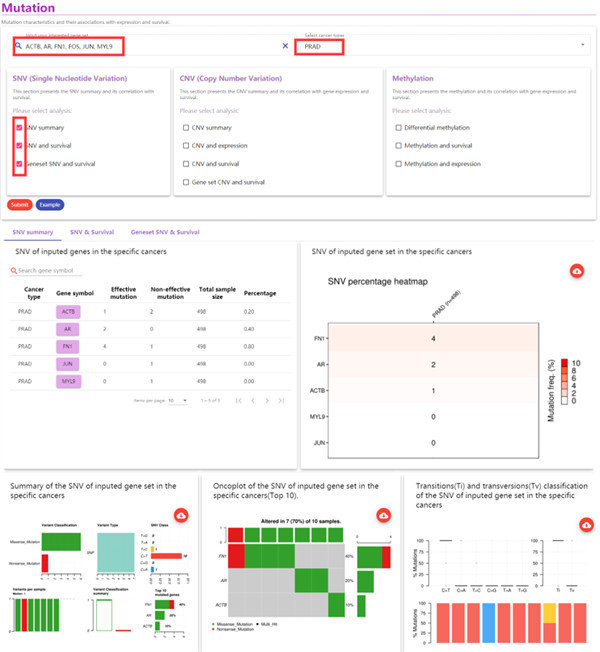
图1.来源查询截图
2、CNV(拷贝数变异)部分则统计展示不同类型及其所占的比例、拷贝数变异与生存的关系、拷贝数变异与表达的关系。如图2
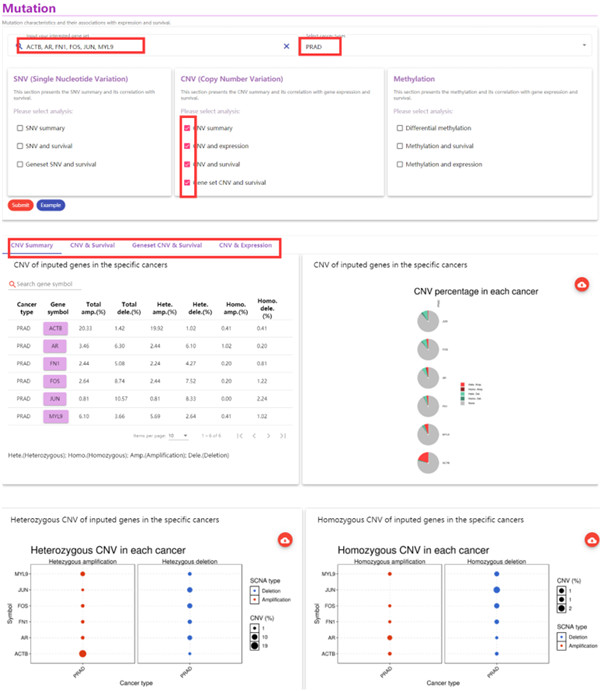
图2.来源查询截图
3、DNA甲基化这部分则给大家展示了基因的差异甲基化、DNA甲基化与生存的关系及DNA甲基化与表达的相关性。如图3
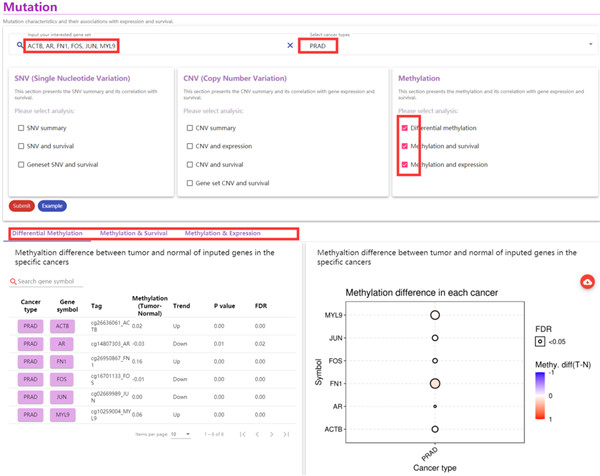
图3. 来源查询截图
方便简单的可以查询到基因水平突变的分布,基因之间是否存在并发或互斥的突变以及其他类型的关联分析,这么好用的挖掘TCGA数据库工具,安利给大家。








